Biyolojik Bir Gerçeklik Olarak Irklar
Bu yazıda, çağdaş sosyal bilimler yazının iddiasının aksine, ırkların biyolojik bir gerçekliğe sahip olduğunu savunacağım.
1. Alt-tür Olarak Irklar
Çok basit bir tanımla, insanlarda “ırk” derken kast ettiğimiz şey, zoolojide “alt-tür”e tekabül eden gruplandırmadır. Tüm taksonomi tanımlarında olduğu gibi alt-tür olma şartları da tartışmalıdır ve bilim insanları farklı şartlar (fizyoloji, morfoloji, psikoloji gibi) kullanabilmektedir. Yine de genel bir kural olarak, bir türe ait iki canlının farklı alt-türler kabul edilebilmesi için aralarında gözle görülebilir biyolojik farklar olması gerektiği söylenebilir. “%75 Kuralı” olarak bilinen kurala göre, bir türe ait bir gruptaki bireylerin %75’i gruptaki tüm bireylerden ayrılabiliyorsa/ayırt edilebiliyorsa, bu bireylerin farklı bir alt-türe ait olduğu söylenebilir. Yine de %75 Kuralı’nı icat etmese dahi popülerleştiren Dean Amadon, %75’in keyfi fakat faydalı bir kesim noktası olduğunu, bu kriterin %99’a da çekilebileceğini söylemektedir (Amadon, 1949). Örneklendirme açısından, Canis lupus türünü (halk adıyla kurtları) ele alalım. Canis lupus, şu anda tanınan 37 alt-türe sahip bir tür. Eğer verili bir Canis lupus popülasyonundaki kurtların önemli bir kısmını diğerlerinden ayırt edip bu popülasyonu ikiye ayırabiliyorsak, iki alt-türe sahibiz demektir. Vücut yapıları, beslenme şekilleri, tüyleri vb. nitelikleriyle birbirinden rahatlıkla ayrılabilen Kuzeybatı Kurdu ve Tundra Kurdu, bu nedenle iki ayrı alt-tür oluşturur (Canis lupus occidentalis ve Canis lupus albus).
Burada alt-tür (veya ırk) için önemli olan kriter, grupla arasında sistematik farklılık bulunmasıdır. Bu farklılıkları ölçmenin birden fazla yolu vardır. Örneğin Tek Nükleotid Polimorfizmi veya otomozal mikrouydular kullanılarak genetik uzaklık veya çeşitlilik ölçümü yapılabileceği gibi, morfolojik (vücut ve organ yapısı) farklılıklar üzerinden de ölçüm ve gruplandırma yapılabilir. Michael Woodley (2009), bazı memeliler için otomozal mikrouydular acılığıyla ölçülen genetik farklılıkları karşılaştırır. Woodley (2009)’dan uyarladığım tabloda “Ho” ile gösterilen “gözlemlenen heterozigotluk”, verili tür içindeki genetik çeşitliliği ifade ediyor, yüksek heterozigotluk, daha yüksek genetik çeşitliliği işaret etmekte.
Tablodan görülebileceği gibi, Homo sapiens sapiens (insan) diğer memelilerin hemen hepsinden yüksek bir genetik çeşitliliğe sahipken “tanınan” bir alt-türü yok. Bize yakın heterozigotluğa sahip şempanzelerin 4, bizden düşük pumaların 6 alt-türü tanınıyor. Burada teorik bir şerh düşmek gerek: Çeşitliliğin yüksekliği veya düşüklüğü kendi başına alt-türleşmeyi getirmek zorunda değil veya alt-tür sayısını belirlemek zorunda değil. Tür içindeki genetik çeşitlilik düşük olsa bile bu çeşitlilik orta-büyük farklar doğurabilecek az sayıda gen üzerinde olabilir; buna ek olarak, bir popülasyonda çeşitliliğin bireyler açısından yüksek olup bunun gruplaşmaya sebep olacak kadar sistematik olmaması da mümkün. Buna yazının ilerleyen kısımlarında detaylı olarak gireceğiz fakat homo sapiens sapiens için hangi ölçütü alırsak alalım, empirik veriler yüksek çeşitliliğin sistematik grup farklarına yol açtığını gösteriyor. Bu yüzden insanların yüksek heterozigotluğa sahip olup alt-türünün tanınmaması taksonomi açısından “kafa karıştırıcı”.
Genetik çeşitliliği ölçmek için kullanılan bir diğer yöntem, Sabitleşme İndeksi (fixation index) ile Fst olarak genetik uzaklık hesaplama. Birbirinden genetik uzaklığı hesaplanan iki popülasyonda Fst=O ise iki popülasyon arasında genetik fark bulunmazken Fst=1 ise birbirlerinden genetik olarak tamamen farklıdırlar demektir. (Nelis et al., 2012)’ye göre, kıtasal popülasyonlar (Avrupa, Afrika ve Asya) için Fst‘ler şöyle:
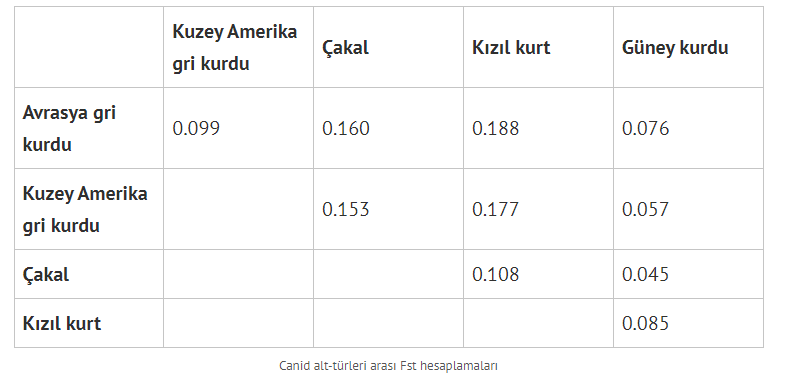
Bu tablo, kurtlar üzerinde yapılan genetik karışım çalışması (vonHoldt et al., 2016) ile karşılaştırınca ilginç bir hâl alıyor:
Avrupa ve Sahra-altı Afrika popülasyonları arasındaki genetik uzaklık ile çakallar ve Kuzey Amerika gri kurdu arasındaki genetik uzaklık eşit (0.153); kimse çakallar ve gri kurtlar arasında yapılacak biyolojik bir ayrımın keyfî/anlamsız olduğunu söylemezken insan grupları arasındaki biyolojik ayrımlara şiddetle karşı çıkılıyor.
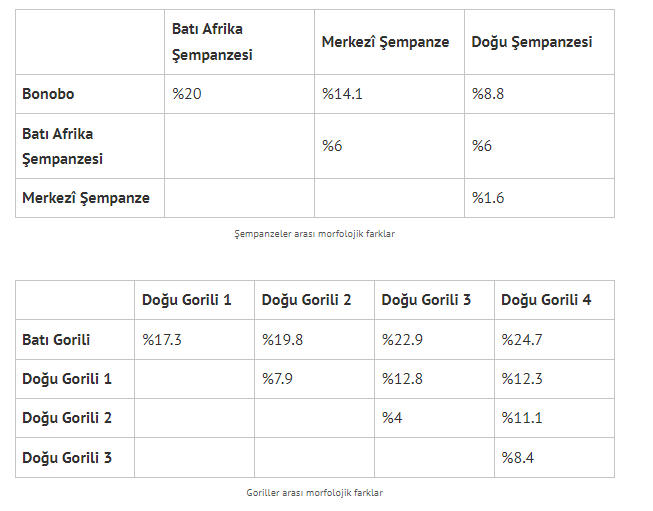
Farklılıklara salt gen düzeyinde değil, morfolojik düzeyde de bakabiliriz. Primat grupları ve evrimi üzerine çalışan antropolog Vincent Sarich’in (Sarich ve Miele, 2004) eserinden uyarladığım tabloyu incelediğimizde, goril ve şempanze alt türleri arasındaki morfolojik farklılıkların, farklı insan grupları arasındaki farklara yakın olduğu görülüyor. [Sarich, goriller için alt tür ismi vermek yerine Batı gorili (G. gorilla) ve dört Doğu gorili popülasyonu diyor. Bahsettiği alt türlerin bilimsel isimlerini araştırırken Doğu gorilleri için genellikle dört değil üç alt tür ayrımı yapıldığını fark ettim, kafa karışıklığına mahal vermemek için Sarich’in taksonomisine ve isimlendirmesine dokunmadan aktardım.]
1.1. Lewontin Safsatası
Biyolojik ırkların gerçekliğine karşı en sık kullanılan argüman, “insanlar için, grup içi farkların gruplar arası farklardan yüksektir, farklılıkların %85’i gruplar arası değil bireyler arasındayken gruplar arası farklar ancak %15’tir, bu nedenle insanlar için alt-türleşme olduğu söylenemez” şeklindedir. Bu argüman, Richard Lewontin’in 1972 tarihli makalesi The apportionment of human diversity‘ye dayanmaktadır. Lewontin, makalesinde, 17 polimorfik loci üzerinden yedi “ırk”ın gen sıklığını analiz etmiş ve “tür çeşitliliğinin %85.4’ünün grup içinde, sadece %15’inden azının gruplar arasında” olduğunu bulmuştur. (Lewontin, 1972)
Bu argümanla ilgili söylenebilecek birkaç şey var. Birincisi, daha önce de kısaca bahsettiğimiz gibi, farkların büyüklüğü kendi başına anlamlı bir metrik değildir. a ve b popülasyonları arasındaki genetik çeşitlilik, diyelim, %5 olsa bile eğer bu %5’lik çeşitlilik orta-büyük derecede farklılık yaratacak genler üzerinde olabilir. Örneğin, Şempanze Dizileme ve Analiz Konsorsiyumu’nun insan ve şempanze genomu karşılaştırmasında, insanlar ve şempanzelerin genom benzerliğinin %96 olduğu gösteriliyor. (Şempanze Dizileme ve Analiz Konsorsiyumu, 2005). İnsan genomunda 3 milyar baz çifti olduğunu düşünürsek, insan ve şempanzeler arasındaki DNA’nın %96’sı aynı olsa bile, %4’lük fark 120 milyon baz çifti farklılığı anlamına gelir, her ne kadar her bir farklılığın kendi başına etkisi görmezden gelinebilir kadar küçük olsa da, bunlar kümülatif biçimde sistematik farklılıklara yol açabilir.
Yakın zamanlı ve ilginç başka bir örnek olarak, Arjantin’in Ibera Milli Parkı’ndaki hemen hemen aynı iki kuş türü, Iberá tohumculları (Sporophila iberaensis) ve sarımsı kahverengi karınlı Capuchino tohumculları (S. hypoxantha) verilebilir. Evrim Ağacı’ndan direkt olarak alıntılarsak:
Arjantin’in Iberá Milli Parkı’nda çalıkuşu boyutlarında, birbirinden neredeyse farksız iki kuş türü, yan yana yaşayıp, günlerini aynı çeşit tohum aramakla ve aynı çeşit yerlerde yuva yapmakla geçiriyor. Hatta bu iki tür, çifteleşerek yavru bile oluşturabilir!
Fakat bu çiftleşme, artık normal koşullarda gerçekleşemiyor. Hem de evrimsel biyologların keşfettiğine bakılırsa çiftleşmenin gerçekleşmeme nedeni, oldukça “önemsiz” gibi görünen özellikler: Karın bölgesindeki hafif renk farklılıkları ve ötme biçimindeki ufak farklılıklar, bu kuşları jenerasyonlar boyunca çiftleşmekten alıkoymak için yeterli olmuş gibi görünüyor.
Bilim insanlarının buluşu, bu kuşların genetik açıdan büyük bir benzerlik paylaşmasına rağmen nasıl olup da iki farklı tür olabildiğini açıklamaya yardımcı olmakta.
…
İki türün daha derinlemesine bir genetik karşılaştırması, onları birbirinden ayıran, toplam 12 genden oluşan üç gen bölgesini açığa çıkardı.
Diğer hayvanlarda da görebildiğimiz gibi, çok ufak farklılıklar, sadece alt-türleşmeyi değil türleşmeyi dahi ortaya çıkarabiliyor.
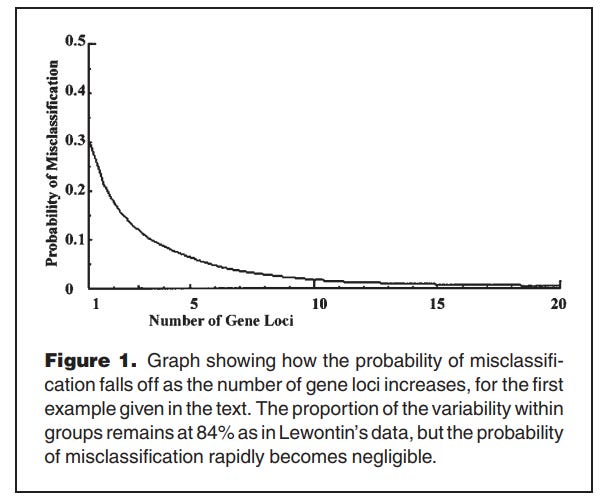
İkincisi, A.W.F. Edwards’ın Human genetic diversity: Lewontin’s fallacy makalesinde gösterdiği gibi, Lewontin, “istatistiksel bir safsata” yapıyor. Edwards, şöyle diyor: “[Lewontin’in bulduğu] bu sonuçlar, veriyi analiz ederken, verinin aynı lokusları karşılaştırarak yapılan analizin ötesinde bir bilgi içermediği varsayımını içeren eski bir istatistiksel safsataya dayanıyor. Genetik verilerinin ‘taksonomik önemi’, çoğu zaman farklı lociler arasındaki korelasyonlardan doğar, zira bunlar istikrarlı bir sınıflandırmayı ortaya çıkarabilecek bilgileri içerebilir.” (Edwards, 2003). Baz alınan lokus sayısı arttıkça grupiçi ve gruplararası genetik çeşitlilik oranları değişmese dahi, istatistiksel dağılımların doğası gereği, sınıflandırmada yanlışlık ihtimali düşüyor. Yani biz her halükarda genetik yapıya bakarak taksonomi yapabiliyoruz, %15 sayısının tartışmada bir önemi olmaması gerekiyor.
Dahası, biz bunu empirik olarak da doğrulayabiliyoruz. (Witherspoon et al., 2007), 3 temel kıta popülasyonu (Avrupalılar, Doğu Asyalılar, Sahra-altı Afrikalılar) ve 5 spesifik popülasyon için (Hintliler, Amerikan Yerlileri, Yeni Gineliler, Afrikan Amerikanlar ve Hispanik-Latinler) Lewontin’i test ediyor. Kullanılan loci sayısı birkaç bin olduğunda hata payı kalmıyor, farklı popülasyonlara ait bireyler birbirlerinden kesin bir biçimde ayrılabiliyor: “Ortak polimorfizmlerde büyük sayıların gücü en açık biçimde Avrupa, Doğu Asya ve Sahra-altı Afrika popülasyonlarının karşılaştırıldığı mikrodizin veri tabanında görülüyor. Benzeşmezlik fraksiyonu davranışı, 1000 polimorfizmde sıfıra yakınsıyor. Bu, yeterli sayıda loci baz alındığında, bu popülasyon gruplarına ait bireylerin, genetik olarak her zaman kendi gruplarının üyelerine benzer olacağını gösteriyor.” (Witherspoon et al, 2007)
2. Kaç ırk var?
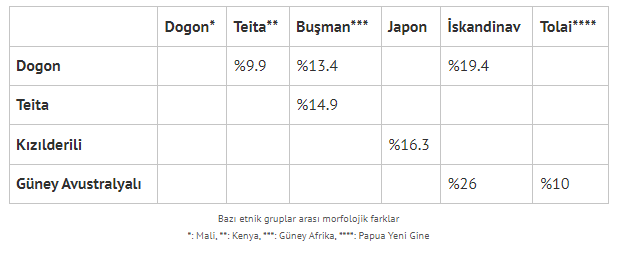
Elimizdeki tüm veriler, insanların, birbirlerinden biyolojik olarak ayrılabilir popülasyonlara -yani alttürlere/ırklara- sahip olduğuna işaret ediyor. Peki, kaç ırk var? Bu soru, ne kadar kaba veya ince bir ayrım yapmak istediğinize göre değişiyor. Homo Sapiens üyeleri, yaklaşık 50 bin yıl önce Afrika’dan ayrıldı ve Avrupa ve Doğu Asya temel olmak üzere farklı kıtalara yayılıp birbirlerinden izole biçimde yaşadılar, evrim hızının son 40 bin senede insanlık tarihinin geri kalanına nazaran 100 kattan fazla arttığını göz önüne alırsak, kıtalar üzerinden ayrım yapıp bu sorunun cevabının “en az 3” olduğunu söyleyebiliriz. Buna (K=3) Modeli diyelim. Buna göre, insan alt-türleri: Caucasoid (Beyaz/Avrupalı), Mongoloid (Asyalı) ve Negroid (Siyahi/Sahra-altı Afrikalı). Bu, ırk tasnifinin en “kaba” biçimi denebilir fakat sistematik farklılıkları burada çok açık biçimde seçebiliyoruz. (Dutton, 2020)’den uyarladığım tabloda bu farkların bazıları görülebilir. Her ne kadar burada sadece 10’a yakın değişken aktarsam da bu üç ırk arasında orta-yüksek derecede farka sahip 40’tan fazla fizyolojik ve psikolojik nitelik mevcut, bunların tamamı için (Dutton, 2020)’ye başvurulabilir.
Fakat (K= 3) Modeli bugün pek sık kullanılan bir model değil zira genetik ölçümler, popülasyonları genellikle 5 ila 9 arasında büyük genetik kümeye ayırıyor. Örneğin, (Li et al., 2008), 938 birey ve 650 bin Tek Nükleotid Poliformizi’ni kullanarak bireyleri, biyolojik kökene göre 7 ırka/popülasyona ayırıyor. Buna (K= 7) Modeli diyebiliriz.
Benzer şekilde, (Xing et al., 2010) da 850 birey ve 46 bin TNP ile aynı kümelenmeyi tekrarlıyor.
Luigi Luca Cavalli-Sforza, magnum opusu The History and Geography of Human Genes (İnsan Genlerinin Tarihi ve Coğrafyası) kitabında, popülasyonların birbirine genetik uzaklıklarına dayanarak 9 kümelenmeli bir ağaç çıkarıyor. (Cavalli-Sforza, Menozzi ve Piazza, 1994) Bunlar: Afrikalılar (Buşmanlar ve Sahraaltı Afrikalılar), Avrupalılar, Kızılderililer, Arktik Asyalılar, Kuzeydoğu Asyalılar, Güneydoğu Asyalılar, Pasifik Adalılar, Yeni Gine ve Avustralya Aborijinleri.
Bunların yanında Nicholas Wade, A Troublesome Inheritance‘ta kolayca ayrılabilir 5 adet popülasyonun/ırkın olduğunu söylüyor: Afrikalılar, Uzak Asyalılar, Avrupalılar, Kızılderililer, Avustralya ve Yeni Gine Aborijinleri. Wade’in (K=5) Modeli de makûliyeti ve pratikliği nedeniyle sıkça kullanılan modellerden.
Bu modeller arasında kıta popülasyonlarını ne kadar ince ayırdıkları dışında belirgin bir fark yok, birbirlerini dışlayan tasnifler değiller.
3. Sonuç ve Bu Neden Önemli?
Bu yazıda, sosyal bilimlerde mevcut “konsensus”un aksine ırkların biyolojik bir gerçeklik olduğunu göstermeye çalıştım. Irkların gerçek olup olmaması ve bunu tartışmak önemli:
Eğer ırklar biyolojik bir gerçeklikse, o halde dünyayı açıklamak için kullanılması gereken değişkenlerden biri olmalıdır. Amerikan Antropoloji Derneği, 1998’de yayınladığı “Irk Üzerine Bildiri”de şöyle diyor: “20. yüzyılın sonunda şunu anlamış bulunmaktayız ki insan kültürel davranışı öğrenilmiştir, doğum anından itibaren bebeklere koşullandırılmıştır ve her zaman değişime tabidir. Hiçbir insan doğuştan kültüre veya dile sahip değildir. Mizacımız, eğilimlerimiz ve kişiliklerimiz, genetik yatkınlıklara bakılmaksızın, ‘kültür’ diye adlandırdığımız anlamlar ve değerler kümesi içinde gelişir. Bebek ve erken çocukluk öğrenim ve davranış çalışmaları, kültürün bizi şekillendirdiği gerçeğinin kanıtıdır. Bütün insanların tüm kültürel davranışları öğrenmeye muktedir olduğu temel bir antropoloji bilgisidir… Sıradan insanların her kültür içinde başarılı ve işlevsel olma kapasitesine dair bildiklerimize dayanarak, günümüzdeki sözde ‘ırk’ grupları arasındaki eşitsizliklerin biyolojik miraslarının değil, tarihsel ve çağdaş toplumsal, iktisadi, eğitimsel ve siyasi koşullarının ürünü olduğu sonucuna varıyoruz.”
Fakat eğer ırklar arası farklar biyolojik birer gerçekse ve bu gerçekler verili bir fenomenle ilişkili olabilecekse, bilimsellik o fenomeni açıklamak için bu değişkeni de test etmemizi ve eğer istatistiksel olarak anlamlıysa açıklamamıza dahil etmemizi gerektirir. Amerikan Antropoloji Derneği’nin bildirisi bu açıdan önemli, zira günümüzde sosyal bilimlerin ve akademinin biyolojik açıklamalara bakışını -düşmanlığını- özetliyor. Çok uzun süredir genel olarak akademinin ve sosyal bilimcilerin duruşu hem ırkların gerçekliğini hem de önemini inkâr etmek, bunun sözünü dahi edenleri dışlamak, şeytanlaştırmak, fiziksel ve psikolojik şiddete maruz bırakmak, baskı altına almak için çabalamak, mobbing yapmak yönünde. Bunun bilimsel değil ideolojik-aktivist bir duruş olduğu apaçık. Irkların bir değişken olarak dikkate alınması bu yüzden de önemli: İdeologluğun bilimi tamamen yutmasına müsaade etmemek için. Biyoloji tek başına her şeyi açıklamaya yetmez, fakat onu tamamen dışarıda bırakmak dünyayı anlama çabasının önünde büyük bir engeldir.
Kaynakça
Amadon, D. (1949). The seventy-five per cent rule for subspecies. The Condor, 51(6), 250–258. https://doi.org/10.2307/1364805
Cavalli-Sforza, L. L., Menozzi, P., & Piazza, A. (1994). The history and geography of human genes. Princeton University Press. shf. 79-80
Dutton, E. (2020). Making sense of race. Washington Summit Publishers.
Edwards, A. W. F. (2003). Human genetic diversity: Lewontin’s fallacy. BioEssays, 25(8), 798–801. https://doi.org/10.1002/bies.10315
Lewontin, R. C. (1972). The apportionment of human diversity. Evolutionary Biology, 381–398. https://doi.org/10.1007/978-1-4684-9063-3_14
Li, J. Z., Absher, D. M., Tang, H., Southwick, A. M., Casto, A. M., Ramachandran, S., Cann, H. M., Barsh, G. S., Feldman, M., Cavalli-Sforza, L. L., & Myers, R. M. (2008). Worldwide human relationships inferred from genome-wide patterns of variation. Science, 319(5866), 1100–1104. https://doi.org/10.1126/science.1153717
Miele, F., & Sarich, V. (2004). Race: The reality of human differences. Westview Press. shf. 170-172
Nelis, M., Esko, T., Mägi, R., Zimprich, F., Zimprich, A., Toncheva, D., Karachanak, S., Piskáčková, T., Balaščák, I., Peltonen, L., Jakkula, E., Rehnström, K., Lathrop, M., Heath, S., Galan, P., Schreiber, S., Meitinger, T., Pfeufer, A., Wichmann, H.-E., … Metspalu, A. (2009). Genetic structure of Europeans: A view from the north–east. PLoS ONE, 4(5). https://doi.org/10.1371/journal.pone.0005472
Şempanze Dizileme ve Analiz Konsorsiyumu. (2005). Initial sequence of the chimpanzee genome and comparison with the human genome. Nature, 437(7055), 69–87. https://doi.org/10.1038/nature04072
vonHoldt, B. M., Cahill, J. A., Fan, Z., Gronau, I., Robinson, J., Pollinger, J. P., Shapiro, B., Wall, J., & Wayne, R. K. (2016). Whole-genome sequence analysis shows that two endemic species of North American wolf are admixtures of the Coyote and Gray Wolf. Science Advances, 2(7). https://doi.org/10.1126/sciadv.1501714
Witherspoon, D. J., Wooding, S., Rogers, A. R., Marchani, E. E., Watkins, W. S., Batzer, M. A., & Jorde, L. B. (2007). Genetic similarities within and between human populations. Genetics, 176(1), 351–359. https://doi.org/10.1534/genetics.106.067355
Woodley, M. A. (2009). Is homo sapiens polytypic? human taxonomic diversity and its implications. Medical Hypotheses, 74(1), 195–201. https://doi.org/10.1016/j.mehy.2009.07.046
Xing, J., Watkins, W. S., Shlien, A., Walker, E., Huff, C. D., Witherspoon, D. J., Zhang, Y., Simonson, T. S., Weiss, R. B., Schiffman, J. D., Malkin, D., Woodward, S. R., & Jorde, L. B. (2010). Toward a more uniform sampling of human genetic diversity: A survey of worldwide populations by high-density genotyping. Genomics, 96(4), 199–210. https://doi.org/10.1016/j.ygeno.2010.07.004